使用BLAST的三個提示(和兩個技巧)
基本局部對齊搜索工具(BLAST)算法是通過國家生物技術信息中心(NCBI)提供的一套免費在線資源的核心。雖然大多數研究人員都知道BLAST是一個序列對齊工具,但NCBI的BLAST套件提供了更多!我將深入介紹如何使用這些資源來定位基因中的單核苷酸多態性(SNPs);用Primer-BLAST設計底漆;驗證引物目標。
提示一:如何找到snp
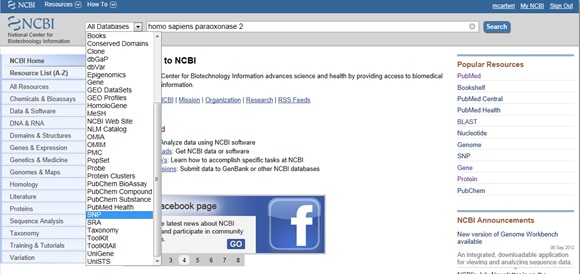
鑒於snp在疾病和研究中的重要性,NCBI提供了工具來整理基因的報告snp。要找到snp,從NCBI開始主頁然後在搜索欄中輸入你感興趣的基因。選擇單核苷酸多態性從所有數據庫搜索欄左側的下拉菜單,如下圖所示:
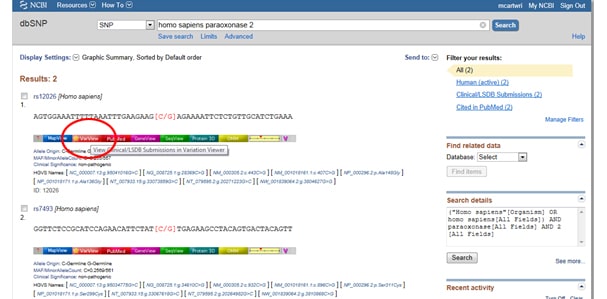
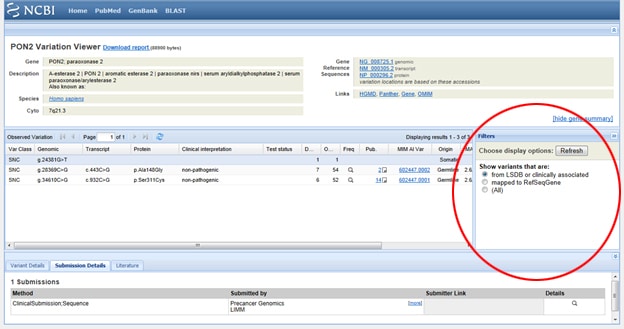
技巧之一:需要過濾結果,以便您隻查看臨床相關的結果?去那個標著過濾器在蘇格蘭民族黨名單的右邊觀察到的變化.選擇過濾器選項後,一定要點擊刷新按鈕。
技巧二:如何設計引語
NCBI提供Primer-BLAST用於根據查詢序列自動設計引物。要開始設計引物,請訪問爆炸的主頁然後向下滾動到Primer-BLAST選擇下專門的爆炸.通過剪切粘貼的方式輸入你的目標序列,或者,如果它在NCBI的數據庫中列出,作為登錄號。我將在下麵介紹一些自定義選項,但在這一點上,您可以生成引物,而無需進行任何額外的自定義!
範圍:在輸入序列的方框右側,您可以指定從序列開始的精確範圍(編號為5 '到3 '),用於設計正向和反向引物的靶標。
使用我自己的正向引物(5 ' ->3 '正鏈):如果你已經設計了你的引物,並希望引物- blast提供一些分析(例如T米)。
PCR產品尺寸:此處設置PCR產物可接受的長度範圍。
返回引物的數量:這設置要考慮的引物候選集的首選數量。注意,這並不是一個保證,特別是當你的參數太嚴格或無意義的時候(例如,你在PCR產品尺寸不會超過500bp,但會低於範圍您隻需考慮間距大於1 KB的引物)。
底漆熔化溫度:這允許您指定T米(要快速了解融化溫度,請查看我們的提示qPCR而且常規PCR引物設計)。
外顯子結跨度:如果你想排除基因組DNA(其中外顯子被非編碼內含子分割),那麼設置為引物必須跨越外顯子-外顯子結.
特異性檢查:除非你希望Primer-BLAST返回偏離目標的引物(一般不推薦!),保持選中並指定生物你們的樣品來自哪裏數據庫來使用,這取決於你的目標是mRNA還是gDNA等等。通過啟用特異性檢查,Primer-BLAST將排除可能放大目標序列之外的東西的引物。
剪接變體處理:如果你選擇這個選項-隻有當你工作的mRNA序列可行-然後Primer-BLAST將不排除可能擴增的引物對多個你目標的mRNA剪接變體。然而,這並不意味著它將提供包含所有已知拚接變體的引物對!你隻是放鬆了你的目標標準。
一旦您輸入了您的序列並根據需要自定義,向下滾動到頁麵底部,在檢查之後使用新的圖形視圖,點擊把引物.這將返回一個地圖,顯示所建議的引物對將在哪裏放大你的目標,以及對引物的分析:它們的長度、精確位置、各自的Tm、GC%和反映自我互補的得分(0.00表示沒有預測的互補)。
提示三:如何預測引物目標
你怎麼檢查你的引物是否擊中了目標?去Primer-BLAST.在查詢框中,輸入您的正向引物(5 '到3 ')。現在輸入一行20個N,將引物分離成單獨的、不重疊的對齊。在N之後,輸入反向引物(也是5 '到3 '),如下所示:
一旦你得到你的結果,檢查它們的某些組合。如果你的前引物在前鏈上對齊(注釋鏈+ / +)而且你的反向引物對齊到相同的命中,但在反向鏈上(鏈+ / -),那麼你的引物可能會放大這種衝擊。
技巧2:你的結果是否包括不太可能汙染你的PCR樣本的東西,如橄欖狒狒和尼安德特人?如果你研究人體樣本或者鼠標樣本,確保你在下麵指定了數據庫.或者,也可以排除特定的物種。
參考資料和其他資源:
爆炸技巧。2007。NCBI。< http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips >
常見問題。NCBI BLAST的幫助。
BLAST序列分析工具。2003。
DW山。使用基本本地對齊搜索工具。2004。冷泉港協議。
惠勒D和Bhagwat M. BLAST快速入門。2007.胡瑪納出版社公司。
3評論
留下你的評論
你必須登錄發表評論。
我有金黃色葡萄球菌的引物序列我在NCBI上進行BLAST但沒有匹配是否有其他BLAST工具來確認這些序列
Blast不是檢查引物特異性的好工具。對於這些非常短的序列,局部對齊算法工作得不是很好。另外一個問題是你隻能搜索公共基因組。MFEprimer或其他類似的軟件工作得更好,它也可以安裝和運行在本地的安撫基因組。
我同意。這篇文章的標題具有誤導性,因為它實際上是關於使用NCBI BLAST,而不是BLAST本身。BLAST是如此之大,比NCBI的web界麵更強大!同樣,我的機體不在NCBI,所以這無關緊要。另一個檢查引物特異性的好方法是ipcress。